النبات

مواضيع عامة في علم النبات

الجذور - السيقان - الأوراق

النباتات الوعائية واللاوعائية

البذور (مغطاة البذور - عاريات البذور)

الطحالب

النباتات الطبية


الحيوان

مواضيع عامة في علم الحيوان

علم التشريح

التنوع الإحيائي

البايلوجيا الخلوية


الأحياء المجهرية

البكتيريا

الفطريات

الطفيليات

الفايروسات


علم الأمراض

الاورام

الامراض الوراثية

الامراض المناعية

الامراض المدارية

اضطرابات الدورة الدموية

مواضيع عامة في علم الامراض

الحشرات


التقانة الإحيائية

مواضيع عامة في التقانة الإحيائية


التقنية الحيوية المكروبية

التقنية الحيوية والميكروبات

الفعاليات الحيوية

وراثة الاحياء المجهرية

تصنيف الاحياء المجهرية

الاحياء المجهرية في الطبيعة

أيض الاجهاد

التقنية الحيوية والبيئة

التقنية الحيوية والطب

التقنية الحيوية والزراعة

التقنية الحيوية والصناعة

التقنية الحيوية والطاقة

البحار والطحالب الصغيرة

عزل البروتين

هندسة الجينات


التقنية الحياتية النانوية

مفاهيم التقنية الحيوية النانوية

التراكيب النانوية والمجاهر المستخدمة في رؤيتها

تصنيع وتخليق المواد النانوية

تطبيقات التقنية النانوية والحيوية النانوية

الرقائق والمتحسسات الحيوية

المصفوفات المجهرية وحاسوب الدنا

اللقاحات

البيئة والتلوث


علم الأجنة

اعضاء التكاثر وتشكل الاعراس

الاخصاب

التشطر

العصيبة وتشكل الجسيدات

تشكل اللواحق الجنينية

تكون المعيدة وظهور الطبقات الجنينية

مقدمة لعلم الاجنة


الأحياء الجزيئي

مواضيع عامة في الاحياء الجزيئي


علم وظائف الأعضاء


الغدد

مواضيع عامة في الغدد

الغدد الصم و هرموناتها

الجسم تحت السريري

الغدة النخامية

الغدة الكظرية

الغدة التناسلية

الغدة الدرقية والجار الدرقية

الغدة البنكرياسية

الغدة الصنوبرية

مواضيع عامة في علم وظائف الاعضاء

الخلية الحيوانية

الجهاز العصبي

أعضاء الحس

الجهاز العضلي

السوائل الجسمية

الجهاز الدوري والليمف

الجهاز التنفسي

الجهاز الهضمي

الجهاز البولي


المضادات الحيوية

مواضيع عامة في المضادات الحيوية

مضادات البكتيريا

مضادات الفطريات

مضادات الطفيليات

مضادات الفايروسات

علم الخلية

الوراثة

الأحياء العامة

المناعة

التحليلات المرضية

الكيمياء الحيوية

مواضيع متنوعة أخرى

الانزيمات
EGF Motif
المؤلف:
I. D. Campbell and P. Bork
المصدر:
Curr. Opin. Struct. Biol. 3, 385–392
الجزء والصفحة:
28-4-2016
2506
EGF Motif
The EGF motif is a common structural domain found in many protein structures, especially those of proteins associated with blood clotting, fibrinolysis, neural development, and cell adhesion. The motif is characterized as a protein module of ~ 45 residues having six conserved cysteine residues ) designated CI through CVI) that form three internal disulfide bonds with the connectivity CI-III, CII-IV and CV-VI. The motif was first found in epidermal growth factor (EGF), hence the name EGF motif or EGF-like domain. Although found as a single unit in EGF and transforming growth factor a, the EGF domain is often present as a repeating unit in much larger proteins. For example, the protein fibrillin has over 30 EGF domains. Furthermore, EGF motifs in proteins are frequently present in combination with repeating units of other modules such as the Kringle domain.
The sequence of the EGF motif is often recognizable from just the primary structure of a protein. The consensus sequence is X–X–X–X–Cys–X(2–7)–Cys–X(1–4)–(Gly/Ala)–X–Cys–X(1–13)–t–t–a–X–Cys–X–Cys–X–X–Gly–a–X(1–6)–Gly–X–X–Cys–X, where X is any amino acid (the number in brackets defining a variable number of X residues), a is an aromatic residue, and t is a nonhydrophobic residue (1). The tertiary structure of the EGF motif is also highly conserved and can be described as having a two-stranded antiparallel b-sheet scaffold upon which the three disulfide bonds are positioned (Fig. 1). The disulfide bonds link the N-terminal and C-terminal loop regions to the core. Binding of calcium ions to some classes of EGF domain further stabilizes the N-terminal region and is thought to cause the formation of long helical arrangements of multiple EGF motifs (2); this property may account for their proposed function of mediating protein–protein interactions.
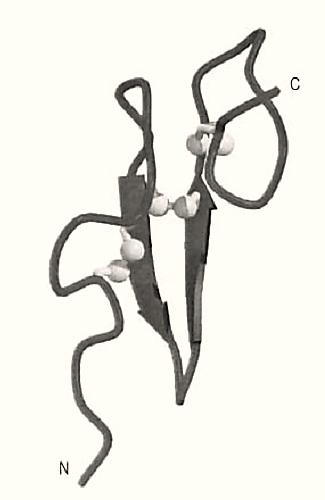
Figure 1. Schematic representation of the backbone structure of an EGF domain (2). b-Strands are shown as arrows, and the three disulfide bonds are shown in light gray, with the sulfur atoms depicted as spheres. The N- and C-termini are labeled. This figure was generated using Molscript (3) and Raster3D (4, 5).
References
1. I. D. Campbell and P. Bork (1993) Curr. Opin. Struct. Biol. 3, 385–392.
2. Z. Rao, P. Handford, M. Mayhew, V. Knott, G. G. Brownlee, and D. Stuart (1995) Cell 82, 131–141.
3. P. J. Kraulis (1991) J. Appl. Crystallogr. 24, 946–950.
4. E. A. Merritt and M. E. P. Murphy (1994) Acta Crystallogr. D50, 869–873.
5. D. J. Bacon and W. F. Anderson (1988) J. Mol. Graphics 6, 219–222.















 قسم الشؤون الفكرية يصدر مجموعة قصصية بعنوان (قلوب بلا مأوى)
قسم الشؤون الفكرية يصدر مجموعة قصصية بعنوان (قلوب بلا مأوى) قسم الشؤون الفكرية يصدر مجموعة قصصية بعنوان (قلوب بلا مأوى)
قسم الشؤون الفكرية يصدر مجموعة قصصية بعنوان (قلوب بلا مأوى) قسم الشؤون الفكرية يصدر كتاب (سر الرضا) ضمن سلسلة (نمط الحياة)
قسم الشؤون الفكرية يصدر كتاب (سر الرضا) ضمن سلسلة (نمط الحياة)